반응형
공분산 (Covariance)
- 두 변수 X, Y가 있을 때,
- 둘이 함께 증가하거나 감소 → 양의 공분산
- 한쪽이 증가할 때 다른 쪽이 감소 → 음의 공분산
- 서로 관련 없음 → 0에 가까움
공분산 행렬
- 변수가 여러 개일 때(예: X₁, X₂, X₃ …), 각 변수 쌍의 공분산을 모두 모아 만든 정사각 행렬
- 대각선 = 각 변수의 분산
- 비대각 = 변수들 사이의 공분산
- Cov(Xᵢ, Xⱼ) = Cov(Xⱼ, Xᵢ) → 대칭행렬)
- Positive semi-definite (행렬이 어떤 방향으로도 음수가 되는 제곱 길이를 만들지 않는 성질)
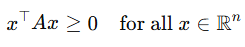
→ 모든 벡터 x에 대해 절대 음수가 되지 않는 성질

PCA
- 공분산이 큰 방향 = 데이터가 가장 퍼져 있는 축
→ 주성분을 찾음
LDA
- 클래스별 공분산 행렬이 동일하다고 가정
Gaussian Mixture Model
- 각 클러스터마다 다른 공분산 행렬을 가짐 (full, diagonal 등 옵션)
다변량 정규분포
- 분산 대신 공분산 행렬로 형태가 결정됨
* cov() 함수로 공분산을 계산할 수 있다.
import pandas as pd
import numpy as np
# 예제 데이터 생성
np.random.seed(0)
X = np.random.normal(0, 1, 100)
Y = 2*X + np.random.normal(0, 1, 100)
# 데이터프레임 구성
df = pd.DataFrame({
'X': X,
'Y': Y
})
# 공분산 행렬 계산
cov_matrix = df.cov()
cov_matrix
공분산 분포 예시
import numpy as np
import matplotlib.pyplot as plt
# Parameters for the grid
n_rows = 10
n_cols = 10
samples = 300
mean = [0, 0]
var_y = 1.0
# Var(X) values (rows): from small to large
var_x_list = np.linspace(0.2, 4.0, n_rows)
# Correlation values (cols): from negative to positive
corr_list = np.linspace(-0.9, 0.9, n_cols)
fig, axes = plt.subplots(n_rows, n_cols, figsize=(10, 10), sharex=False, sharey=False)
for i, var_x in enumerate(var_x_list):
for j, corr in enumerate(corr_list):
cov = corr * np.sqrt(var_x * var_y) # ensures valid covariance
cov_matrix = [[var_x, cov],
[cov, var_y]]
data = np.random.multivariate_normal(mean, cov_matrix, samples)
ax = axes[i, j]
ax.scatter(data[:, 0], data[:, 1], s=8)
# compact titles to avoid clutter
ax.set_title(f"VarX={var_x:.2f}\nρ={corr:.2f}", fontsize=7)
ax.tick_params(axis='both', which='major', labelsize=6)
ax.set_xlim(-4*np.sqrt(var_x), 4*np.sqrt(var_x))
ax.set_ylim(-4*np.sqrt(var_y), 4*np.sqrt(var_y))
plt.suptitle("10×10 grid: rows=Var(X) (small→large), cols=Corr(X,Y) (neg→pos)", fontsize=12)
plt.tight_layout(rect=[0, 0, 1, 0.96])
plt.show()

반응형

댓글