SW 역량테스트 합격하기 A형 강의 오픈!! (인프런 바로가기)

먼저 좌표가 무제한이기 때문에, 어느 정도의 크기로 MAP을 선언할지 생각해야 한다.
K가 최대 300번, 총 300번 시뮬레이션해야하고, 가장 빨리 증식하는 세포는 생명력 수치가 1이다.
따라서 2시간에 1번씩 4방향으로 계속 퍼지므로, 300번동안 최대 150 만큼 MAP이 커진다.
어느 정도 여유를 두기 위해 MAP은 500 x 500 중심점(OFFSET)은 250으로 두었다.
#define MAX (500)
#define OFFSET (250)
int T, N, M, K;
int MAP[MAX][MAX];
MAP은 줄기세포의 생명력 수치를 표시하기 위해 사용한다.
줄기세포를 퍼지게 하는 것은 줄기세포 구조체 CELL의 배열이다. 아래와 같이 구성한다.
typedef struct st
{
int r;
int c;
int life;
int time;
}CELL;먼저 자신의 좌표를 가지고 있어야하므로 (r, c)를 필요로 한다.
life는 생명력 수치이고, time은 증식하기까지 걸리는 시간을 life에서 감소하기 위해 선언하였다.
즉, time이 0이 되면 4방향 증식을 시작하면 된다.
문제가 복잡하기 때문에 필요한 CELL의 종류도 다양하다.
cell : 시뮬레이션을 위한 cell
liveCell : cell에서 탐색 후, 남아 있는 cell. 즉, time이 0 이상인 cell (비활성 상태)
spreadCell : cell의 time이 0이 되고, 4방향으로 증식해야 하는 cell
deadCell : time이 0이 된 cell이며, 문제에서 제시한 "활성 상태"를 위한 cell (활성 상태)
CELL cell[MAX * MAX];
CELL liveCell[MAX * MAX];
CELL spreadCell[MAX * MAX];
CELL deadCell[MAX * MAX];
int ccnt, lcnt, scnt, dcnt;
deadCell은 cell에서 활성 상태가 된 cell 그 자체이며, spreadCell은 4방향으로 퍼져나간 cell이 된다.
K 시간이 지난 후, 활성 상태의 세포도 정답에 포함하기 때문에 deadCell(곧 죽을 cell)을 따로 관리한다.
deadCell에 저장된 cell들은 life를 감소시켜서 life가 0이 되면 최종적으로 죽은 것으로 판별한다.
즉, 마지막에 출력하는 값은 liveCell의 수 + deadCell의 수가 된다.
input에서는 MAP의 (r + OFFSET, c + OFFSET) 부터 입력을 받는다.
cell의 ccnt를 초기화하고, MAP에 0이 아닌 값인 경우는 cell 배열에 좌표와 생명력, 그리고 time = 생명력으로 받는다.
void input()
{
scanf("%d %d %d", &N, &M, &K);
for (int r = 0; r < MAX; r++)
for (int c = 0; c < MAX; c++)
MAP[r][c] = 0;
ccnt = 0;
for (int r = 0; r < N; r++)
{
for (int c = 0; c < M; c++)
{
int life;
scanf("%d", &life);
MAP[r + OFFSET][c + OFFSET] = life;
if (life)
{
cell[ccnt].r = r + OFFSET;
cell[ccnt].c = c + OFFSET;
cell[ccnt].life = life;
cell[ccnt++].time = life;
}
}
}
}시뮬레이션은 아래와 같이 진행한다.
1-1) cell을 보고 time이 0이 아닌 경우, liveCell에 cell 추가.
1-2) time이 0인 경우는 deadCell에 추가.
1-3) time이 0인 경우, 4방향 탐색해서, MAP에 빈칸인 경우 spreadCell에 Cell 추가.
2) deadCell의 life 감소, life가 남아있으면 다시 deadCell에 추가.
3-1) MAP이 빈칸이라면, spreadCell의 life를 MAP에 표시.
3-2) MAP이 빈칸이 아니고, 자신의 life가 더 크다면 MAP 갱신.
3-3) MAP이 빈칸이 아니고, 자신의 life가 같거나 작다면, spreadCell의 time을 0으로 변경
4) 3) 과정 종료 후, spreadCell이 0이 아닌 경우만 liveCell에 추가.
5) 다음 1) 과정을 다시 하기 위해 liveCell을 cell에 추가.
이 과정을 K번 반복한다.
1) 과정은 아래와 같다.
liveCell과 spreadCell은 cell을 보고 저장하기 때문에, lcnt와 scnt의 초기화가 필요하다.
indexMAP은 3) 과정에서 설명한다.
spreadCell에 추가할 때, MAP에 이미 cell이 있는 경우는 무시해야한다.
이 과정은 spreadCell이 동시에 한 곳을 차지하는 것과는 다르다.
lcnt = scnt = 0;
int indexMAP[MAX][MAX] = { 0 }; /* 3) 과정에서 필요 */
for (int i = 0; i < ccnt; i++)
{
/* 1-1) cell을 보고 time이 0이 아닌 경우, liveCell에 cell 추가. */
if (cell[i].time)
{
cell[i].time--;
liveCell[lcnt++] = cell[i];
}
else
{
/* 1-2) time이 0인 경우는 deadCell에 추가. */
deadCell[dcnt] = cell[i];
dcnt++;
/* 1-3) time이 0인 경우, 4방향 탐색해서, MAP에 빈칸인 경우 spreadCell에 Cell 추가. */
for (int dir = 0; dir < 4; dir++)
{
int nr, nc;
nr = cell[i].r + dr[dir];
nc = cell[i].c + dc[dir];
if (MAP[nr][nc]) continue;
spreadCell[scnt].r = nr;
spreadCell[scnt].c = nc;
spreadCell[scnt].life = cell[i].life;
spreadCell[scnt++].time = cell[i].life;
}
}
}
4방향 탐색을 위해 미리 dr, dc 배열을 선언해두자.
/* 순서대로 왼쪽, 위, 오른쪽, 아래 */
int dr[] = { 0, -1, 0, 1 };
int dc[] = { -1, 0, 1, 0 };2) 과정은 아래와 같다. deadCell의 life를 1 감소하고, 여전히 life가 있는 경우만 deadCell에 저장한다.
/* 2) deadCell의 life 감소, life가 남아있으면 다시 deadCell에 추가. */
int ddcnt = 0;
for (int i = 0; i < dcnt; i++)
{
deadCell[i].life--;
if (deadCell[i].life)
deadCell[ddcnt++] = deadCell[i];
}
dcnt = ddcnt;따로 배열을 추가하지 않고, deadCell에 활성 상태의 cell을 다시 저장한다.
과정을 그림으로 보면 아래와 같다. 초기 상태는 dcnt = 5이고 life가 1, 0, 3, 0, 1이라고 하자.
그리고 ddcnt = 0으로 초기화한다.

i = 0 에서 life가 있으므로, deadCell에 그대로 저장하고, ddcnt를 1 증가한다.

i = 1 에서 life가 0이므로, 무시한다.

i = 2 에서 life가 3이므로, deadCell에 저장하고 ddcnt를 1 증가한다.
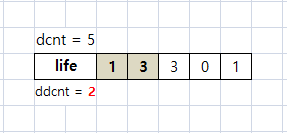
i = 3에서 life가 0이므로 무시한다.

i = 4에서 life가 1이므로, deadCell에 저장하고 ddcnt를 1 증가한다.

마지막으로 dcnt = ddcnt로 변경하면 i = 3, i = 4에 어떤 값이 있든, 접근할 일이 없게 된다.
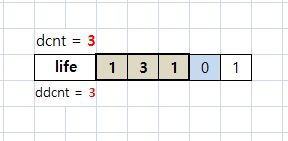
life가 남은 deadCell의 수는 K 시간이 남은 이후의 dcnt가 된다.
3) 과정은 indexMAP을 이용하여 spreadCell의 좌표를 기억해야 한다.
MAP에 이미 좌표가 있는 경우는 앞의 spreadCell이 먼저 빈 칸을 차지한 경우이다.
자신의 life가 더 크면 이 값을 바꿔줘야 하지만, 앞의 spreadCell을 지울 방법이 없다.
따라서 indexMAP에 spreadCell의 i 값, index를 기억하여 spreadCell[indexMAP[nr][nc]]의 time을 0으로 만든다.
time이 0이 된 값을 나중에 한 번 더, spreadCell을 돌면서 liveCell에 제외하면 된다.
for (int i = 0; i < scnt;i++)
{
int nr, nc;
nr = spreadCell[i].r;
nc = spreadCell[i].c;
/* 3-1) MAP이 빈칸이라면, spreadCell의 life를 MAP에 표시. */
if (MAP[nr][nc] == 0)
{
MAP[nr][nc] = spreadCell[i].life;
indexMAP[nr][nc] = i;
}
/* 3-2) MAP이 빈칸이 아니고, 자신의 life가 더 크다면 MAP 갱신. */
else if (MAP[nr][nc] < spreadCell[i].life)
{
spreadCell[indexMAP[nr][nc]].time = 0;
MAP[nr][nc] = spreadCell[i].life;
indexMAP[nr][nc] = i;
}
/* 3-3) MAP이 빈칸이 아니고, 자신의 life가 같거나 작다면, spreadCell의 time을 0으로 변경 */
else
spreadCell[i].time = 0;
}
indexMAP은 이전에 초기화하였으나, MAX가 너무 크면 stack이 커져서 실행이 잘 안되는 경우가 발생한다.
따라서 MAX를 넉넉하게 잡되, 함수의 stack 사이즈보다 작게 선언하는 것이 좋다.
(보통 주어진 문제에서 함수 내에서는 1MB 이하로 메모리를 사용한다.)

만약 MAX를 더 넉넉하게 잡고 싶다면, indexMAP을 전역 변수로 선언하고, 매 time마다 0으로 초기화 하면 된다.
마지막으로 4) ~ 5)의 과정을 보자.
3)에서 life가 작아서 쓸모없어진 spreadCell의 time은 0이 되었으므로, time이 1 이상인 cell을 liveCell로 옮긴다.
1) 과정을 다시 하기 위해 liveCell을 cell로 옮기면 한 과정이 모두 끝난다.
/* 4) 3) 과정 종료 후, spreadCell이 0이 아닌 경우만 liveCell에 추가. */
for (int i = 0; i < scnt; i++)
if (spreadCell[i].time) liveCell[lcnt++] = spreadCell[i];
/* 5) 다음 1) 과정을 다시 하기 위해 liveCell을 cell에 추가. */
for (int i = 0; i < lcnt; i++) cell[i] = liveCell[i];
ccnt = lcnt;마지막으로 lcnt와 dcnt를 더하면 줄기 세포(비활성 상태 + 활성 상태)의 총 개수가 된다.
최종 코드는 아래를 참고하자.
#include <stdio.h>
#define MAX (500)
#define OFFSET (250)
int T, N, M, K;
int MAP[MAX][MAX];
typedef struct st
{
int r;
int c;
int life;
int time;
}CELL;
CELL cell[MAX * MAX];
CELL liveCell[MAX * MAX];
CELL spreadCell[MAX * MAX];
CELL deadCell[MAX * MAX];
int ccnt, lcnt, scnt, dcnt;
void input()
{
scanf("%d %d %d", &N, &M, &K);
for (int r = 0; r < MAX; r++)
for (int c = 0; c < MAX; c++)
MAP[r][c] = 0;
ccnt = 0;
for (int r = 0; r < N; r++)
{
for (int c = 0; c < M; c++)
{
int life;
scanf("%d", &life);
MAP[r + OFFSET][c + OFFSET] = life;
if (life)
{
cell[ccnt].r = r + OFFSET;
cell[ccnt].c = c + OFFSET;
cell[ccnt].life = life;
cell[ccnt++].time = life;
}
}
}
}
void output()
{
for (int r = OFFSET - 5; r < OFFSET + N + 5; r++)
{
for (int c = OFFSET - 5; c < OFFSET + M + 5; c++)
printf("%d ", MAP[r][c]);
putchar('\n');
}
putchar('\n');
}
/* 순서대로 왼쪽, 위, 오른쪽, 아래 */
int dr[] = { 0, -1, 0, 1 };
int dc[] = { -1, 0, 1, 0 };
int main(void)
{
scanf("%d", &T);
for (int tc = 1; tc <= T; tc++)
{
input();
for (int time = 0; time < K; time++)
{
lcnt = scnt = 0;
int indexMAP[MAX][MAX] = { 0 }; /* 3) 과정에서 필요 */
for (int i = 0; i < ccnt; i++)
{
/* 1-1) cell을 보고 time이 0이 아닌 경우, liveCell에 cell 추가. */
if (cell[i].time)
{
cell[i].time--;
liveCell[lcnt++] = cell[i];
}
else
{
/* 1-2) time이 0인 경우는 deadCell에 추가. */
deadCell[dcnt] = cell[i];
dcnt++;
/* 1-3) time이 0인 경우, 4방향 탐색해서, MAP에 빈칸인 경우 spreadCell에 Cell 추가. */
for (int dir = 0; dir < 4; dir++)
{
int nr, nc;
nr = cell[i].r + dr[dir];
nc = cell[i].c + dc[dir];
if (MAP[nr][nc]) continue;
spreadCell[scnt].r = nr;
spreadCell[scnt].c = nc;
spreadCell[scnt].life = cell[i].life;
spreadCell[scnt++].time = cell[i].life;
}
}
}
/* 2) deadCell의 life 감소, life가 남아있으면 다시 deadCell에 추가. */
int ddcnt = 0;
for (int i = 0; i < dcnt; i++)
{
deadCell[i].life--;
if (deadCell[i].life)
deadCell[ddcnt++] = deadCell[i];
}
dcnt = ddcnt;
for (int i = 0; i < scnt;i++)
{
int nr, nc;
nr = spreadCell[i].r;
nc = spreadCell[i].c;
/* 3-1) MAP이 빈칸이라면, spreadCell의 life를 MAP에 표시. */
if (MAP[nr][nc] == 0)
{
MAP[nr][nc] = spreadCell[i].life;
indexMAP[nr][nc] = i;
}
/* 3-2) MAP이 빈칸이 아니고, 자신의 life가 더 크다면 MAP 갱신. */
else if (MAP[nr][nc] < spreadCell[i].life)
{
spreadCell[indexMAP[nr][nc]].time = 0;
MAP[nr][nc] = spreadCell[i].life;
indexMAP[nr][nc] = i;
}
/* 3-3) MAP이 빈칸이 아니고, 자신의 life가 같거나 작다면, spreadCell의 time을 0으로 변경 */
else
spreadCell[i].time = 0;
}
/* 4) 3) 과정 종료 후, spreadCell이 0이 아닌 경우만 liveCell에 추가. */
for (int i = 0; i < scnt; i++)
if (spreadCell[i].time) liveCell[lcnt++] = spreadCell[i];
/* 5) 다음 1) 과정을 다시 하기 위해 liveCell을 cell에 추가. */
for (int i = 0; i < lcnt; i++) cell[i] = liveCell[i];
ccnt = lcnt;
}
printf("#%d %d\n", tc, lcnt + dcnt);
}
return 0;
}'알고리즘 > [ADV] 삼성 SW Expert Academy A형' 카테고리의 다른 글
| SWEA 5648 : 원자 소멸 시뮬레이션 (모의 SW 역량테스트) (0) | 2021.04.26 |
|---|---|
| SWEA 5650 : 핀볼 게임 (모의 SW 역량테스트) (0) | 2021.04.22 |
| SWEA 5656 : 벽돌 깨기 (모의 SW 역량테스트) (0) | 2021.04.15 |
| SWEA 5658 : 보물상자 비밀번호 (모의 SW 역량테스트) (0) | 2021.04.12 |
| 삼성 A형 샘플 문제 : 프로세서 연결하기 (0) | 2021.04.11 |





댓글